Compreendendo o diagnóstico de APEC por qPCR
O diagnóstico preciso das infecções por APEC (Avian Pathogenic Escherichia coli) emerge como pilar fundamental para mitigar riscos sanitários e econômicos na avicultura. Embora essas infecções, denominadas colibacilose aviária, gerem grandes perdas, em razão da mortalidade elevada, condenação de carcaças e perdas de desempenho, o maior obstáculo reside na distinção entre cepas comensais (AFEC - Avian fecal E. coli) e as verdadeiramente patogênicas (APEC). Este desafio no diagnóstico laboratorial é um dos maiores entraves para estratégias eficazes.
A dificuldade está diretamente relacionada à grande plasticidade genética de E. coli, uma espécie com notável capacidade de adquirir e trocar genes por meio de plasmídeos, bacteriófagos e outros elementos genéticos móveis. Essa variabilidade faz com que a patogenicidade não possa ser inferida apenas com base nas semelhanças ou diferenças entre cepas, como quando se consideram os sorotipos antigênicos – somático (O), flagelar (H) e capsular (K). Assim, cepas de um mesmo sorotipo podem ser altamente patogênicas ou completamente comensais. A ausência de correlação direta entre características fenotípicas e patogenicidade reflete a intensa dinâmica de recombinação e mobilidade genética da espécie.
Apesar dessa complexidade, estudos genômicos nas últimas décadas permitiram definir com maior precisão o patótipo APEC, mostrando que a maioria dos isolados patogênicos se concentra em um cluster genômico conservado, portador de um conjunto específicos de genes que os distinguem dos isolados comensais classificados como AFEC (2).
Diante dessa complexidade, o diagnóstico de APEC passou a ser tratado como presuntivo e probabilístico, integrando biologia molecular e análise estatística. A ideia central é que a presença combinada de determinados genes preditores de patogenicidade aumenta a probabilidade de uma cepa ser patogênica. Assim, a detecção simultânea de múltiplos genes preditores por qPCR define a probabilidade de relação entre o perfil genético e o comportamento patogênico, e não a associação de fatores de virulência isolados (3).
|
Genes preditores de patogenicidade
Genes de virulência
|
Nesse contexto, o estudo de Johnson et al. (2008) (3) representou um avanço no diagnóstico molecular de APEC. Os autores analisaram centenas de isolados de E. coli provenientes de aves doentes e sadias, identificando um conjunto de genes cuja presença estava fortemente associada ao comportamento patogênico. Por meio de uma análise estatística multivariada, foram definidos cinco genes preditores de patogenicidade — iss, iutA, ompT, hlyF e iroN — que, quando detectados em conjunto por PCR, demonstraram alta acurácia na identificação de isolados APEC. Esses genes mostraram boa correspondência com os patótipos de APEC e foram capazes de discriminar isolados patogênicos de não patogênicos (AFEC) com desempenho comparável à genotipagem de 46 genes de virulência. No estudo, os isolados classificados como APEC apresentaram, em média, 4,0 dentre 5 genes preditores, enquanto os isolados AFEC apresentaram, em média, 1,3 genes. Assim, concluiu-se que a presença de quatro ou mais desses genes indica alta probabilidade de o isolado ser APEC.
A Simbios, ao longo de vários anos, reuniu um banco de dados robusto de amostras (n=520) de E. coli isoladas de diferentes origens. Foram analisadas amostras por qPCR multiplex para iss, iutA, ompT, hlyF e iroN, com dois parâmetros principais avaliados:
Tabela 1. Percentual de APEC e AFEC identificadas — conforme a classificação de Johnson et al. (2008), onde a presença de 4 ou 5 alvos foi determinada como APEC:
| Categoria | n | % |
|---|---|---|
| APEC | 280 | 54% |
| AFEC | 240 | 46% |
Gráfico 1. Distribuição do número de genes preditores de patogenicidade detectados por amostra de E. coli aviária.
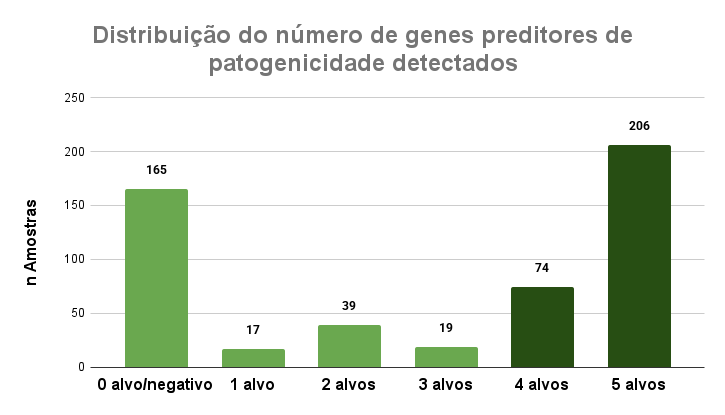
O conjunto de dados revelou que a contagem do número de alvos não segue distribuição normal (centrada em valor intermediário), mas sim uma concentração marcante de resultados em dois extremos (bimodal). Não seguir distribuição normal indica não aleatoriedade na presença dos genes preditores de virulência. A distribuição bimodal reforça a capacidade discriminatória entre AFEC e APEC do modelo proposto por Johnson et al. (2008), confirmando que a abordagem baseada em múltiplos genes preditores é diagnosticamente discriminante e reprodutível.
Referências:
Veja mais:
SERVIÇOS | Diagnóstico molecular para o setor avícola
NewGene FastX: rapidez, simplicidade e eficiência na obtenção de DNA bacteriano